Como pasamos un proceso de 11 horas a 37 minutos
Introducción
Este artículo es muy similar al que Ulises escribió en su momento sobre como pasó un proceso de 5 horas a 5 minutos pero en un contexto diferente ya que no se trata de optimizar el código en NodeJS sino que hablamos de Python aplicado a Ingeniería de datos y en este caso ha sido pasar un proceso de 11 horas a 37 min. Pero mejor dejadme que os pongo en contexto:
Problemas (Winter is coming)
Actualmente disponemos de una herramienta llamada Apache Airflow para gestionar la automatización de procesos. A estos, se les puede asignar mediante un crontab la frecuencia con la que se van a ejecutar y con ello dejar que hagan por su cuenta el trabajo especificado mediante código en Python.
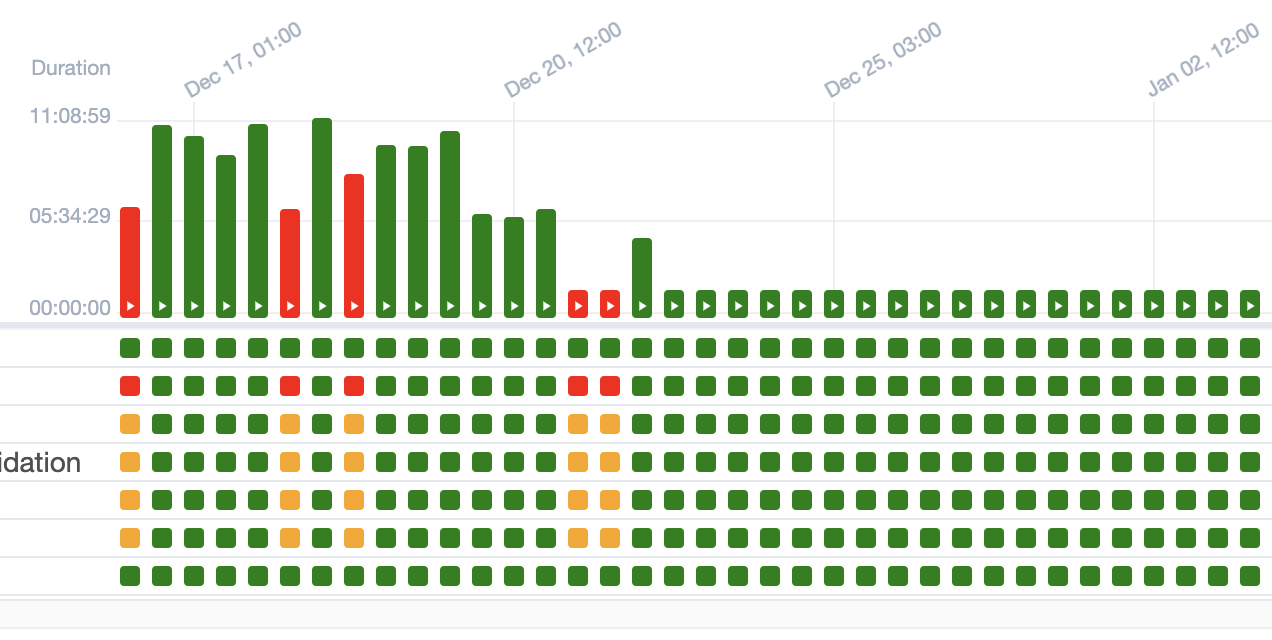
Uno de los muchos procesos que tenemos es el de extracción de datos de proveedores y su posterior tratamiento para que sean adaptados al dominio. Este proceso normalmente tenía una duración media de 5 horas lo cual era mas o menos pasable por varios motivos:
- Es un proceso automático que lo dejamos ejecutando normalmente de madrugada y a primera hora de la mañana ya lo tenemos ejecutado
- Maneja un volumen de datos elevado y todas las operaciones que ejecutamos llevan tiempo
- No es necesario tenerlo al momento por lo que no es crítico para ningún otro equipo
Aquí dejo una captura para que comprueben que es algo que de vez en cuando tiene sus fallos pero que se solucionan con alguna revisión y suele ser algo estable:
Y llegó el invierno
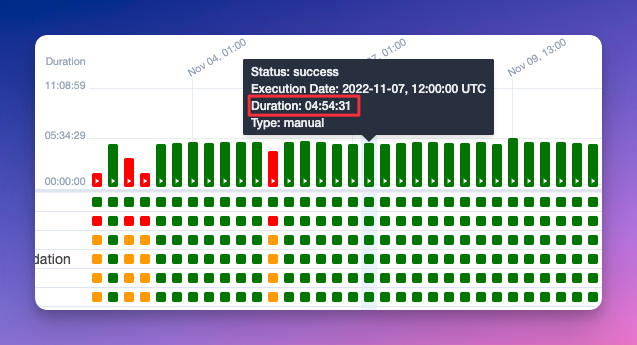
Por un motivo u otro, se añadieron a la cola de procesado unos pocos millones más de datos y eso terminó por incrementar en exceso los tiempos de ejecución. Concretamente se pasó de 5 horas a 11:
Esto ya se estaba yendo de nuestras manos y no era sostenible que un proceso durase casi medio día y que se lanza 2 veces en 24 horas… Hagan sus cuentas.
¿Por qué ocurrió esto?
Es conocido que para tratamiento de Datos se usa una librería por excelencia que es Pandas y funciona muy bien la verdad. Sin embargo, para cantidades absurdas de datos tiene un pésimo rendimiento y ahí nació otra librería que usamos para optimizar grandes volúmenes que es Dask.
En cantidades grandes de datos Dask le pega un repaso interesante a Pandas. Pero hay veces que Dask puede ser un poco dolor de cabeza si no se usa bien y existen muchos libros al respecto con buenas prácticas. Entre ellos les recomiendo Data Science at Scale with Python and Dask.
Entonces suponíamos que algo mal estábamos haciendo con Dask y nos dispusimos a revisar el código.
Misterios misteriosos
Primero revisamos qué hacía nuestro proceso:
Recoge 3 datasets, los junta en 1 y procesa los datos aplicando una lista de prioridad en base al origen de los datos. Lo que llamamos una
provider_rank_list(tengan esto presente porque volveremos aquí)
Los 3 datasets de origen no vienen en 1 único fichero cada uno, sino que están particionados ya que Dask los divide para que le sea más fácil realizar las operaciones. Aquí nos dimos cuenta que las particiones eran minúsculas. Estamos hablando de 1028 particiones de 1-2 Mb cada una… Efectivamente esto mata a Dask y todo lo bueno y óptimo que tiene termina por ser todo lo contrario.
Por lo que una de las mejoras que implementamos fue reducir esas 1028 particiones a ficheros más grandes (pero manteniendo particiones para que Dask haga lo suyo).
Así que aquí viene el super consejito del día:
Si trabajas con Dask, asegúrate de particionar acorde al volumen de datos. No tengas demasiadas particiones pequeñas ni pocas particiones muy grandes. Según la documentación oficial se recomiendan particiones de ~100Mb
No me gustaría dejarles este ejemplo sin un pedacito de código para asegurar que se entiende el proceso:
|
|
¿Por qué fijarle un tamaño a las particiones y no un número fijo de ficheros y que haga Dask todo el trabajo?
Me alegra que te hagas esa pregunta. Dask puede ser un poco dolor de cabeza. Existen 2 formas de particionar: npartitions y partition_size. npartitions fija un número de particiones, ni más ni menos de las que tu le indicas. Por otro lado partition_size fija el tamaño máximo de cada partición y además provoca que el Dask Dataframe se procese, lo cual a priori es negativo porque consume más recursos y tarda más. Pero no estábamos teniendo en cuenta que el procesado se iba a realizar sí o sí, porque para guardarlo se transforma a Pandas (no lo había dicho antes pero Dask está construido sobre Pandas).
TLDR; La repartición estaba funcionando regular y nos leímos mucha documentación para utilizar la opción que mejor nos venía
Y colorín colorado… Este cuento aún no se ha acabado :(
¿Mejoró el rendimiento? Sí. ¿Todo lo que nos gustaría? No. El proceso aún se tiraba unas cuantas horas buenas y seguía siendo inviable. Había que seguir buscando…
Hasta que dimos con una línea de código
|
|
¿Qué hace el set_index? -> Ordena el dataframe en base al parámetro que le indicas. En este caso como queremos filtrar datos en base a proveedores, pues queremos ordenarlos en base a unos numeritos. Por ejemplo:
|
|
Pues esto anteriormente, cuando se implementó nos parecía guay y una idea factible… Pero al final no escalable ni mucho menos. A medida que aumentaba el volumen de datos, peor era el rendimiento de Dask para ordenar.
Seguro que si has llegado hasta aquí, esperarás una solución ultra compleja, casi como una receta mágica… Lamento decirte que la solución no tiene que ver con código sino con planteamiento del problema.
Solución
Como bien se había dicho, el proceso coge 3 datasets y los junta en 1 para procesar los datos en base a una priority_rank_list. Pues se nos ocurrió concatenar (es una operación casi instantánea y que consume muy poco) en base a la lista de prioridad. Así nos olvidamos de ordenar el resultado porque ya nos vendrá ordenado y supondrá mucha menos carga de trabajo. Tan sencillo como eso.
Esto en código no tiene mucho sentido ponerlo pero los resultados hablan por sí solos:
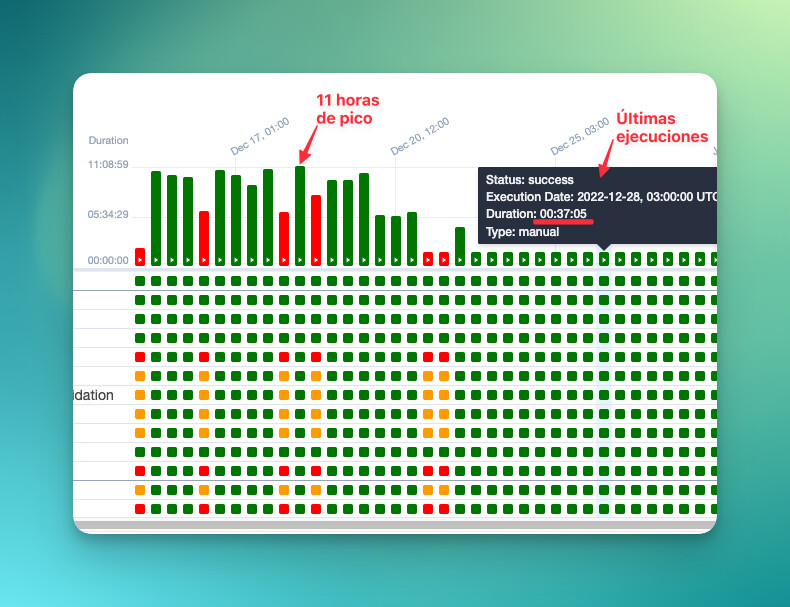
Y por si fuese de interés, este planteamiento lo extendimos a los procesos que preceden. Les dejo una tabla con las mejoras de tiempo:
Antes
| Proceso 1 | Proceso 2 | Proceso 3 | Tiempo total | |
|---|---|---|---|---|
| Proveedor A | ~20 min | ~8h50min | ~11 horas | 20h10min |
| Proveedor B | ~20 min | ~3h30min | ~11 horas | 14h50min |
| Proveedor C | ~10 min | ~7h | ~11 horas | 11h17min |
Después
| Proceso 1 | Proceso 2 | Proceso 3 | Tiempo total | |
|---|---|---|---|---|
| Proveedor A | ~20 min | ~1h | ~1-2h | 3h20min |
| Proveedor B | ~20 min | ~45min | ~1-2h | 3h5min |
| Proveedor C | ~10 min | ~7min | ~1-2h | 2h17min |
Al final del día no solo hemos ahorrado tiempo de ejecución, sino también recursos de los Pods de Kubernetes que ejecutan esto y máquinas de AWS. Lo cual se traduce en menos dinero gastado
Y como bien se suele decir: Una imagen vale más que mil palabras, por lo que les dejo una foto final de como la tormenta se calmó: